认识序列
在程序员的角度看待序列,就是字符串。
我们这里研究的序列主要是指蛋白质序列和核酸序列。其中蛋白质序列是由20个不同的氨基酸排列组合而成。核酸则是由4中不同的碱基排列组合而成,核酸序列又分为DNA序列和RNA序列。
其中最常用的就是第二章中讲过的FASTA格式。
同源性、序列相似性、一致性
同源性是断定。如果两个序列有一个共同的进化祖先,那么它们是同源的。
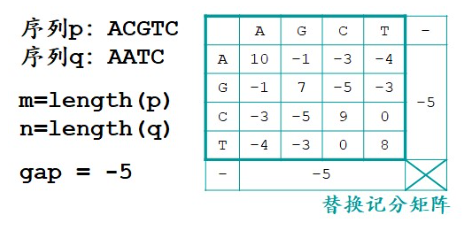
序列的相似性、一致性是定量描述了序列有多相似。一致性是指对应位置上相同的残基数目占总长度的百分比。相似性是对应位置上相似的残基与相同的残基的数目和占总长度的百分比,而相似的评估需要替换记分矩阵。
替换记分矩阵
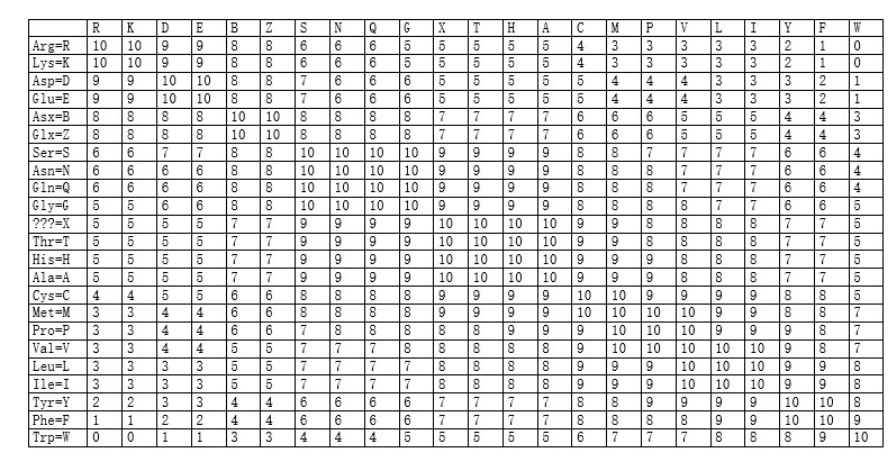
替换记分矩阵是反映残基之间相互替换率的矩阵。描述了残基两两相似的量化关系。例如下图是蛋白质的BLOSUM-62替换记分矩阵。

核酸序列的替换记分矩阵
核酸的碱基包括两类:嘌呤(腺嘌呤A、鸟嘌呤G)类都有两个环;嘧啶(胞嘧啶C、胸腺嘧啶T)类有一个环。 其中环数不变的变化称为转换;环数变化的变化称为颠换。
DNA序列的替换记分矩阵主要有三种:
- 等价矩阵:相同得1,不同得0。
- 转换-颠换矩阵:转换发生的频率远比颠换高。
- BLAST矩阵:经验矩阵。
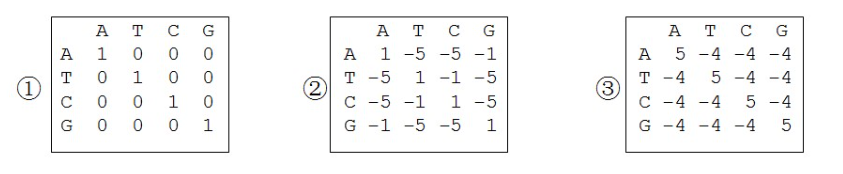
蛋白质序列的替换记分矩阵
- 等价矩阵:相同得1,不同得0。
- PAM矩阵:基于进化原理,由统计方法得到。
如下图是PAM250矩阵。对角线上的数值为匹配氨基酸的得分。其他位置上≥0的得分代表对应的一对氨基酸为相以氨基酸,<0的是不相似的氨基酸。
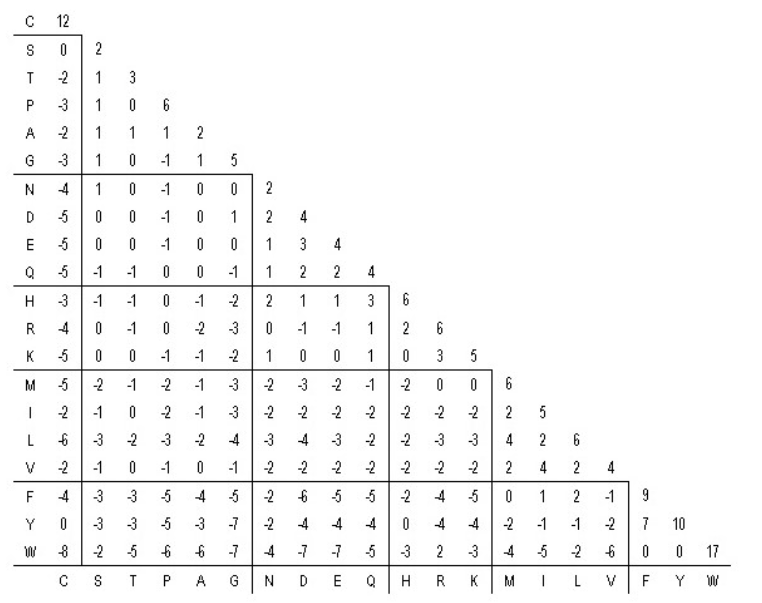
- BLOSUM矩阵:和PAM类似,BLOSUM矩阵的相似性是根据真实数据产生的。

如何选择替换矩阵?
PAM的编号体现的是序列差异性;BLOSUM的编号体现的是序列相似性(直等于)。
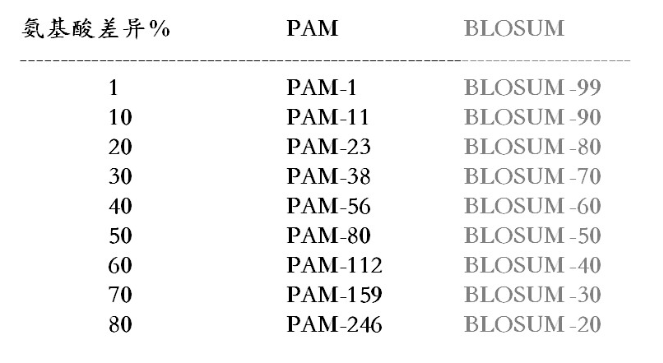
即亲缘关系较近的序列之间的比较,用PAM数小的矩阵或 BLOSUM数大的矩阵;而亲缘关系较远的序列之间的比较,用PAM数大的矩阵或BLOSUM数小的矩阵。
那究竟是APM还是BLOSUM呢?
对亲缘关系较远的,APM矩阵是自乘过来的,会有一定误差,BLOSUM更有优势;而对于亲缘关系较近的,两者的差别不大。
简单来说的话:记住BLOSUM62走遍天下。
除了APM和BLOSUM,还有两个蛋白质的替换记分矩阵。 3. 遗传密码矩阵:通过计算一个氨基酸转换成另一个氨基酸所需要的密码子变化的数目而得到。优点在于计算结果能够直接用于绘制进化树。

- 疏水矩阵:根据氨基酸替换前后疏水性的变化而得到的矩阵。
那么问题又来了,我如何比对两个长度不一致的序列呢?我们先系统了解一下序列比较的方法。
序列比较
打点法
方法理论
该方法理论上能够用纸和笔来完成 : )
我们将要对比的序列分别水平书写和竖直书写。然后相同残基位置打点。如下图所示。
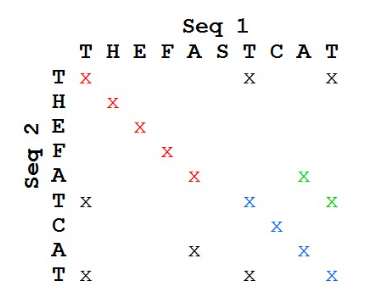
可以发现其中连续对角线及对角线的平行线代表了两条序列的相同的区域。
我们用不相关的两条序列来进行打点:
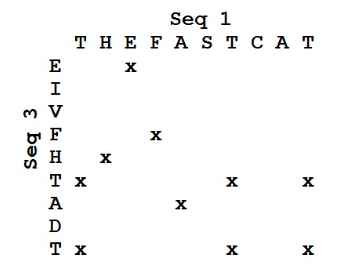
可以发现没有明显的连续的线。
该方法还能够用于查序列中的重复片段,即自己和自己进行打点。其中平行线重复的次数就是重复片段的次数。如下图中的THE。
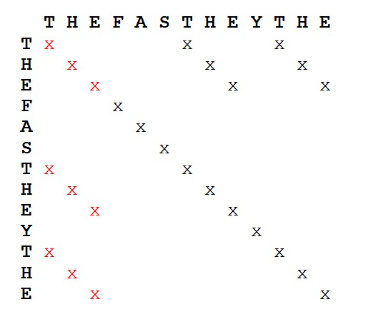
还能够发现序列中的串联重复序列以及重复的次数。只要数出来再半个矩阵中包括主对角线在内的所有等距的平行线个数,就是重复的多次数。并且最短的平行线对应的序列就是重复单元。
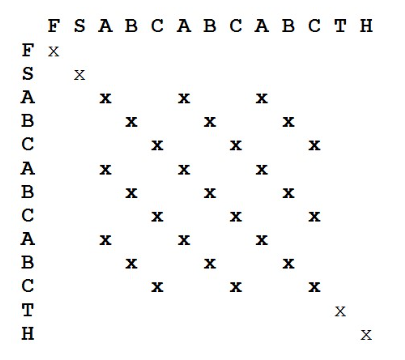
打点软件
序列比对法
序列比对是运用算法找出两个或者多个序列之间产生最大相似度得分的空格插入和序列排列方案。
- 双序列比对:全局比对、局部比对
- 多序列比对
双序列全局比对
经典的全局比对算法是Needleman-Wunsch算法。运用的是动态规划的思想。
今天所有的比对软件的算法都是从这个算法衍生出来的。
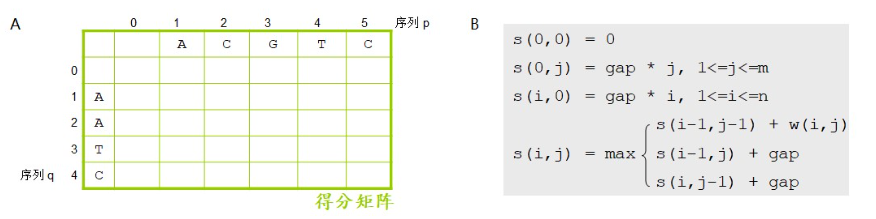
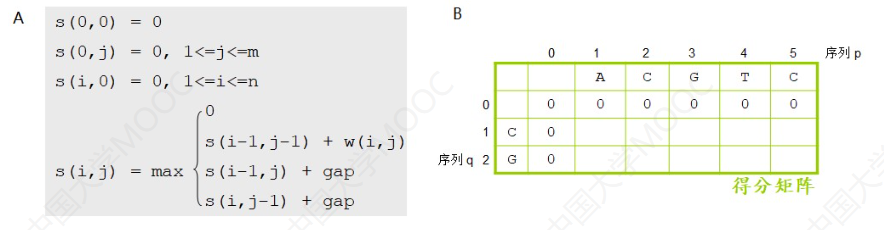
输入:序列p和序列q;替换记分矩阵;空位罚分。
接着我们创建一个得分矩阵,并根据公式把得分矩阵填满。
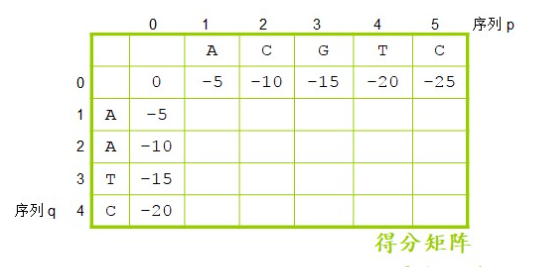
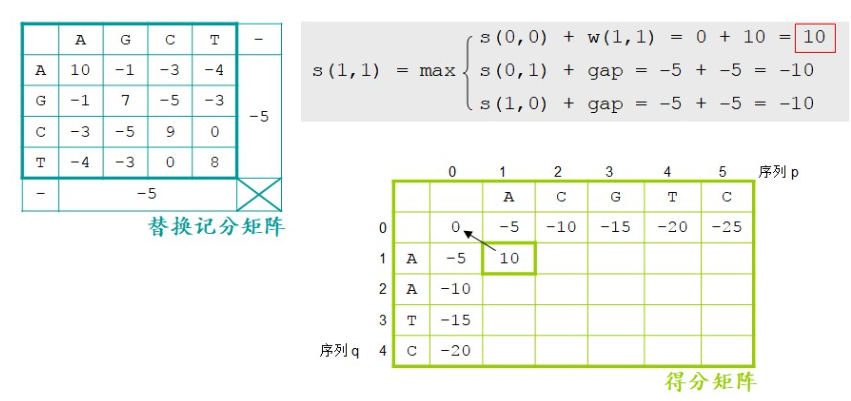
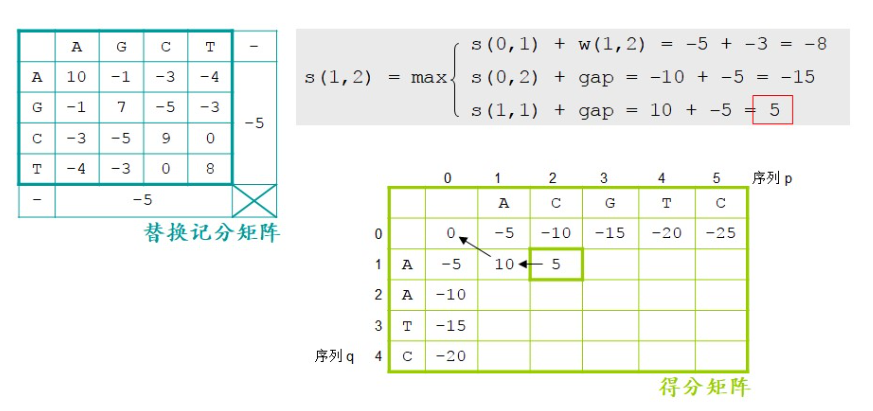
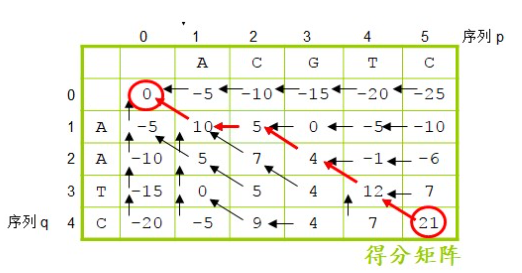
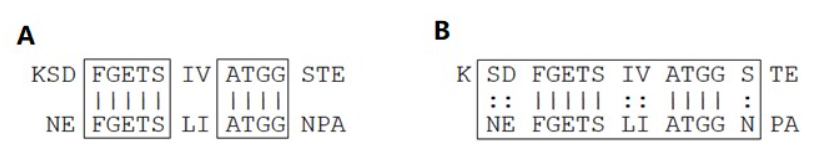
这对计算机专业的学生并不难。填满之后,我们就能回溯出来比对结果。最后回溯的时候,是从左上角开始,如果是斜箭头则是字符对字符,如果是水平箭头或垂直箭头则是字符对空位,箭头指着的序列为空位。
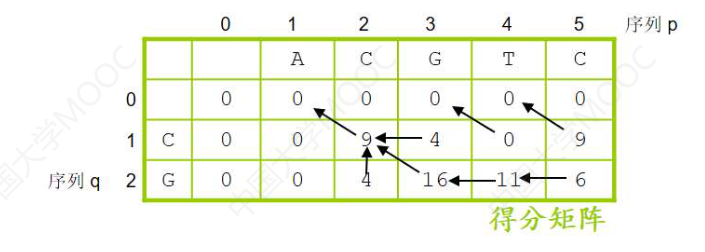
我们看第一个是斜箭头,字母对字母,就是A对A,第二个是水平箭头,字母对空位,箭头指着的序列是空位,也就是C对空位。然后斜箭头G对A,斜箭头T对T,斜箭头C对C,一直写到右下角,全局比对就出现了。

双序列局部比对
局部比对是在全局比对的基础上增加了第四个元素“0”。
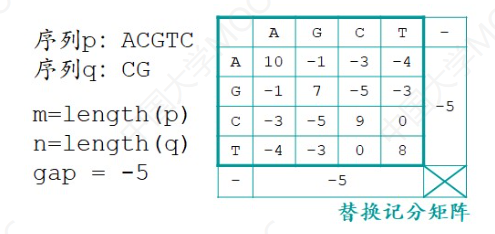
最终结果是:
与全局比对不同的是,局部比对是在矩阵中找最大值,并且从最大值开始回溯。

在线序列比对工具
BLAST
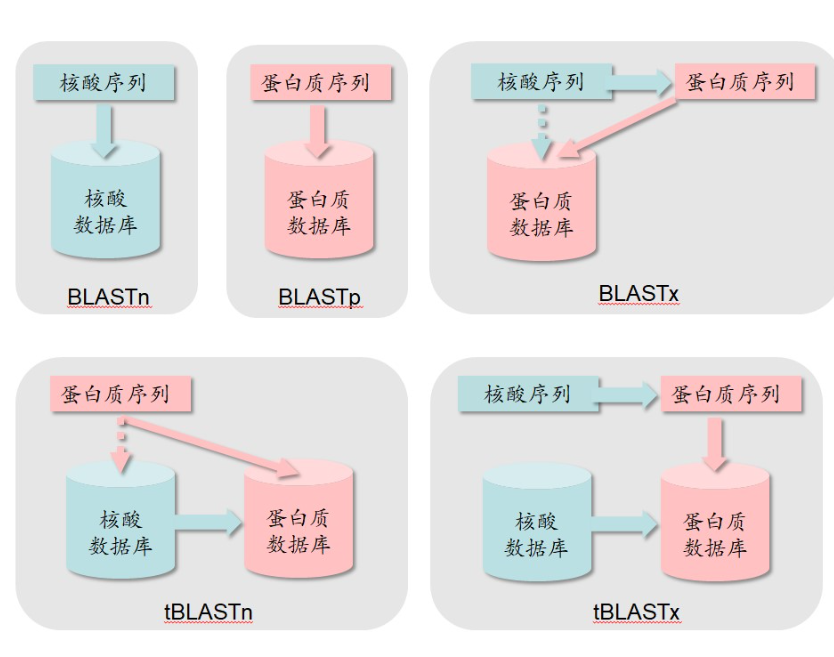
基本局部比对搜索工具。
BLAST是基于片段对进行的搜索,将计算机复杂度降为n。
BLAST是一组工具的统称:
- BLASTp
- BLASTn
- BLASTx
- tBLASTn
- tBLASTx
多序列比对
两条以上的生物序列进行的全局比对就是多序列比对。

通过多序列比对能够:
- 确定某一个位置序列是否属于某一个家族。
- 构建系统发生树,查看进化关系。
- 模式识别,寻找保守片段。
做n序列比对,就是创建了一个n维空间。
在线工具
T-COFFEE Multiple Sequence Alignment Server
Job Dispatcher < EMBL-EBI
寻找保守区域
说了这么多的序列比对方法,其实我们目的就是想要从其中获得序列中重要的位置。即保守位置。
序列标识图,就是序列的logo。用图形的方式依次画出序列比对中各个位置上出现的残基,在该位置上出现的频率越大,字符大小也就越大。
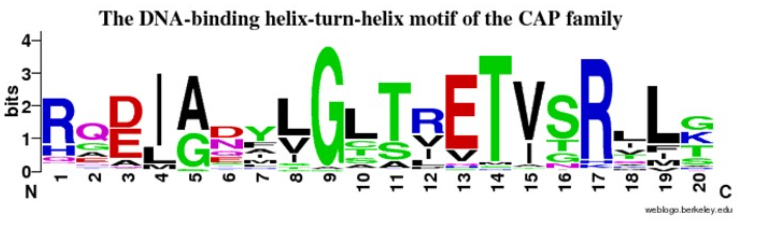
如果某一列非常保守,那么字母高度就高。反之,各种残基都有就没有什么特征。
在线软件:WebLogo 3 - About
Motif基序
Motif是在核酸或蛋白质序列中存在一些有特定模式的序列片段。