生物数据库
由于生物基因序列、蛋白质结构、理化信息等等生物数据的剧增,建立生物数据库对数据进行管理迫在眉睫。
时至今日,世界上的生物数据库的数量已经很难统计了。
著名的学术期刊Nucleic Acids Research有一个生物数据库专刊。有点儿规模的数据库都争相在这里发表。包括Genbank,PDB等等,都在这里发表更新版本。截止2015年底,这个专刊收录的生物数据库累计1685个。当然还有一些在其他刊物发表的小型专项数据库。加上这些,目前世界上得有超过2000个生物数据库。
当然,不是所有数据库都是活的。已发表的数据库中有相当数量的数据库在发表之后就死去了。事实上,数据库不是一个可以放在那里保质期无限长的罐头,他需要专业人员不断的管理、维护和更新。这些工作一旦停止,数据库便立刻失去了生命力。
所以如果将来有一天,你也有了自己开发的数据库,请善待它,不要轻易抛弃他。
数据库的分类
我们可以将生物数据库分成三大类:
- 核酸数据库
- 蛋白质数据库
- 专用数据库
核酸数据库和蛋白质数据库又分为一级和二级。
一级数据库存储的是通过各种科学手段得到的最直接的基础数据。比如测序获得的核酸序列,或者X射线衍射法等获得的蛋白质三维结构。蛋白质的一级数据库还可以再具体分为蛋白质序列数据库和蛋白质结构数据库。
二级数据库是通过对一级数据库的资源进行分析、整理、归纳、注释而构建的具有特殊生物学意义和专门用途的数据库。比如从三大核酸数据库和基因组数据库中提取并加工出的果蝇和蠕虫数据库,再比如根据蛋白质三维结构数据库中的结构信息,分析统计出的蛋白质结构分类数据库CATH和SCOP等。
文献数据库
PubMed:生物医学文献数据库,其中大部分文献来自于
MEDLINE。arXiv:由康奈尔大学维护的一个非营利文献数据库,主要涉及物理、数学、计算机科学、定量生物学、定量金融、统计学、电气工程与系统科学、经济学等领域。
一级核酸数据库
一级核酸数据库主要包括三大核酸数据库和基因组数据库。三大核酸数据库包括NCBI的Genbank,EMBL的ENA和DDBJ,它们共同构成国际核酸序列数据库。
这里以GenBank数据库为例,分别浏览一个原核生物的基因和一个真核生物的基因,并解读其中的信息。
原核生物:基因组小;基因密度高,1000个碱基里就有1个基因;编码区含量高;基因是呈线性分布的;没有内含子。
真核生物:基因组大;基因密度低,比如人,要10万个碱基才有1个基因;编码区含量低;基因是非线性的,因为翻译蛋白质的外显子被内含子分隔开来,真核生物的RNA要经历剪切的过程,剪切后的成熟mRNA才能进行翻译,这是原核生物和真核生物基因的最大区别;
原核生物核酸序列
在GenBank官网上搜索序号X01714,其中Nucleotide数据库就是GenBank数据库。
Title: E. coli dut gene for dUTPase (EC 3.6.1.23) (deoxyuridine 5'-triphosphate nucleotidohydrolase)
LOCUS X01714 1609 bp DNA linear BCT 23-OCT-2008
DEFINITION E. coli dut gene for dUTPase (EC 3.6.1.23) (deoxyuridine
5'-triphosphate nucleotidohydrolase).
ACCESSION X01714
VERSION X01714.1
KEYWORDS dUTPase; unidentified reading frame.
SOURCE Escherichia coli
ORGANISM [Escherichia coli](https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=562)
Bacteria; Pseudomonadati; Pseudomonadota; Gammaproteobacteria;
Enterobacterales; Enterobacteriaceae; Escherichia.
REFERENCE 1 (bases 1 to 1609)
AUTHORS Lundberg,L.G., Thoresson,H.O., Karlstrom,O.H. and Nyman,P.O.
TITLE Nucleotide sequence of the structural gene for dUTPase of
Escherichia coli K-12
JOURNAL EMBO J. 2 (6), 967-971 (1983)
PUBMED [6139280](https://www.ncbi.nlm.nih.gov/pubmed/6139280)
COMMENT Data kindly reviewed (25-NOV-1985) by L. Lundberg.
FEATURES Location/Qualifiers
source 1..1609
/organism="Escherichia coli"
/mol_type="genomic DNA"
/db_xref="taxon:[562](https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=562)" [regulatory](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=286&to=291) 286..291
/regulatory_class="promoter"
/note="-35 region" [regulatory](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=310&to=316) 310..316
/regulatory_class="promoter"
/note="-10 region" [misc_feature](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=322&to=324) 322..324
/note="put. transcription start region" [regulatory](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=330&to=333) 330..333
/regulatory_class="ribosome_binding_site"
/note="put. rRNA binding site" [CDS](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=343&to=798) 343..798
/note="unnamed protein product; dUTP-ase (aa 1-151)"
/codon_start=1
/transl_table=[11](https://www.ncbi.nlm.nih.gov/Taxonomy/Utils/wprintgc.cgi?mode=c#SG11)
/protein_id="[CAA25859.1](https://www.ncbi.nlm.nih.gov/protein/41297)"
/db_xref="GOA:[P06968](https://www.ebi.ac.uk/ego/GProtein?ac=P06968)"
/db_xref="InterPro:[IPR008180](https://www.ebi.ac.uk/interpro/entry/InterPro/IPR008180)"
/db_xref="InterPro:[IPR008181](https://www.ebi.ac.uk/interpro/entry/InterPro/IPR008181)"
/db_xref="PDB:[1DUD](https://www.rcsb.org/structure/1DUD)"
/db_xref="PDB:[1DUP](https://www.rcsb.org/structure/1DUP)"
/db_xref="PDB:[1EU5](https://www.rcsb.org/structure/1EU5)"
/db_xref="PDB:[1EUW](https://www.rcsb.org/structure/1EUW)"
/db_xref="PDB:[1RN8](https://www.rcsb.org/structure/1RN8)"
/db_xref="PDB:[1RNJ](https://www.rcsb.org/structure/1RNJ)"
/db_xref="PDB:[1SEH](https://www.rcsb.org/structure/1SEH)"
/db_xref="PDB:[1SYL](https://www.rcsb.org/structure/1SYL)"
/db_xref="PDB:[2HR6](https://www.rcsb.org/structure/2HR6)"
/db_xref="PDB:[2HRM](https://www.rcsb.org/structure/2HRM)"
/db_xref="UniProtKB/Swiss-Prot:[P06968](https://www.uniprot.org/uniprot/P06968)"
/translation="MKKIDVKILDPRVGKEFPLPTYATSGSAGLDLRACLNDAVELAP
GDTTLVPTGLAIHIADPSLAAMMLPRSGLGHKHGIVLGNLVGLIDSDYQGQLMISVWN
RGQDSFTIQPGERIAQMIFVPVVQAEFNLVEDFDATDRGEGGFGHSGRQ" [misc_feature](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=831&to=851) 831..851
/note="put.stem-loop structure" [repeat_region](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=831&to=838) 831..838
/note="inverted repeat A" [repeat_region](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=844&to=851) 844..851
/note="inverted repeat A'" [misc_feature](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=866&to=893) 866..893
/note="put. stem-loop structure" [repeat_region](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=866&to=872) 866..872
/note="imp. inverted repeat B" [repeat_region](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=888&to=893) 888..893
/note="imp. inverted repeat B'" [regulatory](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=889&to=895) 889..895
/regulatory_class="ribosome_binding_site"
/note="pot. rRNA binding site" [CDS](https://www.ncbi.nlm.nih.gov/nuccore/X01714.1?from=905&to=1540) 905..1540
/note="unnamed protein product; unidentified reading
frame"
/codon_start=1
/transl_table=[11](https://www.ncbi.nlm.nih.gov/Taxonomy/Utils/wprintgc.cgi?mode=c#SG11)
/protein_id="[CAA25860.1](https://www.ncbi.nlm.nih.gov/protein/41298)"
/db_xref="GOA:[P0C093](https://www.ebi.ac.uk/ego/GProtein?ac=P0C093)"
/db_xref="InterPro:[IPR001647](https://www.ebi.ac.uk/interpro/entry/InterPro/IPR001647)"
/db_xref="InterPro:[IPR009057](https://www.ebi.ac.uk/interpro/entry/InterPro/IPR009057)"
/db_xref="InterPro:[IPR011075](https://www.ebi.ac.uk/interpro/entry/InterPro/IPR011075)"
/db_xref="InterPro:[IPR015893](https://www.ebi.ac.uk/interpro/entry/InterPro/IPR015893)"
/db_xref="UniProtKB/Swiss-Prot:[P0C093](https://www.uniprot.org/uniprot/P0C093)"
/translation="MAEKQTAKRNRREEILQSLALMLESSDGSQRITTAKLAASVGVS
EAALYRHFPSKTRMFDSLIEFIEDSLITRINLILKDEKDTTARLRLIVLLLLGFGERN
PGLTRILTGHALMFEQDRLQGRINQLFERIEAQLRQVLREKRMREGEGYTTDETLLAS
QILAFCEGMLSRFVRSEFKYRPTDDFDARWPLIAASCSNMTPDDFSSGEFL"
ORIGIN
1 cagagaaaat caaaaagcag gccacgcagg gtgatgaatt aacaataaaa atggttaaaa
61 accccgatat cgtcgcaggc gttgccgcac taaaagacca tcgaccctac gtcgttggat
121 ttgccgccga aacaaataat gtggaagaat acgcccggca aaaacgtatc cgtaaaaacc
181 ttgatctgat ctgcgcgaac gatgtttccc agccaactca aggatttaac agcgacaaca
241 acgcattaca ccttttctgg caggacggag ataaagtctt accgcttgag cgcaaagagc
301 tccttggcca attattactc gacgagatcg tgacccgtta tgatgaaaaa aatcgacgtt
361 aagattctgg acccgcgcgt tgggaaggaa tttccgctcc cgacttatgc cacctctggc
421 tctgccggac ttgacctgcg tgcctgtctc aacgacgccg tagaactggc tccgggtgac
481 actacgctgg ttccgaccgg gctggcgatt catattgccg atccttcact ggcggcaatg
541 atgctgccgc gctccggatt gggacataag cacggtatcg tgcttggtaa cctggtagga
601 ttgatcgatt ctgactatca gggccagttg atgatttccg tgtggaaccg tggtcaggac
661 agcttcacca ttcaacctgg cgaacgcatc gcccagatga tttttgttcc ggtagtacag
721 gctgaattta atctggtgga agatttcgac gccaccgacc gcggtgaagg cggctttggt
781 cactctggtc gtcagtaaca catacgcatc cgaataacgt cataacatag ccgcaaacat
841 ttcgtttgcg gtcatagcgt gggtgccgcc tggcaagtgc ttattttcag gggtattttg
901 taacatggca gaaaaacaaa ctgcgaaaag gaaccgtcgc gaggaaatac ttcagtctct
961 ggcgctgatg ctggaatcca gcgatggaag ccaacgtatc acgacggcaa aactggccgc
1021 ctctgtcggc gtttccgaag cggcactgta tcgccacttc cccagtaaga cccgcatgtt
1081 cgatagcctg attgagttta tcgaagatag cctgattact cgcatcaacc tgattctgaa
1141 agatgagaaa gacaccacag cgcgcctgcg tctgattgtg ttgctgcttc tcggttttgg
1201 tgagcgtaat cctggcctga cccgcatcct cactggtcat gcgctaatgt ttgaacagga
1261 tcgcctgcaa gggcgcatca accagctgtt cgagcgtatt gaagcgcagc tgcgccaggt
1321 attgcgtgaa aagagaatgc gtgagggtga aggttacacc accgatgaaa ccctgctggc
1381 aagccagatc ctggccttct gtgaaggtat gctgtcacgt tttgtccgca gcgaatttaa
1441 ataccgcccg acggatgatt ttgacgcccg ctggccgcta attgcggcca gttgcagtaa
1501 tatgacgccg gatgactttt catccggcga gtttctttaa acgccaaact cttcgcgata
1561 ggccttaacc gccgccagat gttccgccat ttccggcttc tcttccagg
//
从记录可以知道dUTPase是脱氧尿苷焦磷酸酶,编码他的基因叫dut基因,所属物种是大肠杆菌。
LOCUS:这一行里包括基因座的名字,核酸序列长度,分子的类别,拓扑类型,原核生物的基因拓扑类型都是线性的,最后是更新日期。
其中bp是Base Pair的缩写,代表的是碱基对。
DEFINITION:是这条序列的简短定义,也就是前面看到的标题。
ACCESSION:就是在搜索条中输入的那个数据库编号,也叫做检索号,每条记录的检索号在数据库中是唯一且不变的。
Version :版本号的格式是“检索号点上一个数字”。主要用于识别数据库中一条单一的特定核苷酸序列。在数据库中,如果某条序列发生了改变,即使是单碱基的改变,它的版本号都将增加,而它的Accession也就是检索号保持不变。比如,版本号由U12345.1 变为U12345.2,而检索号依然是U12345。版本号后面还有个GI号。GI号与前面的版本号系统是平行运行的。当一条序列改变后,它将被赋予一个新的GI号,同时它的版本号将增加。
KEYWORDS提供能够大致描述该条目的几个关键词,可用于数据库搜索。
SOURCE:基因序列所属物种的俗名。他下面还有一个子条目,ORGANISM,是对所属物种更详细的定义,包括他的科学分类。
REFERENCE:是基因序列来源的科学文献。有时一条基因序列的不同片段可能来源于不同的文献,那样的话,就会有很多个REFERENCE条目出现。REFERENCE的子条目包括文献的作者、题目和刊物。刊物下面还包括PubMed ID作为其子条目。
COMMENT:是自由撰写的内容,比如致谢,或者是无法归入前面几项的内容。
FEATURES:是非常重要的注释内容,它描述了核酸序列中各个已确定的片段区域,包含很多子条目,比如来源,启动子,核糖体结合位点等等。
source:说明了核酸序列的来源,据此可以容易的分辨出:这条序列是来源于克隆载体还是基因组。可以看到,当前序列来源于大肠杆菌的基因组DNA。
promoter:列出了启动子的位置。细菌有两个启动子区,-35区和-10区。-35区位于第 286个碱基到第291个碱基,-10区位于第310个碱基到第316个碱基。
misc_feature:列出了一些杂项,比如,这条说明了从第322个碱基到第324个碱基是一个推测的,但无实验证实的转录起始位置。
RBS:是核糖体结合位点的位置。
CDS:Coding Segment,编码区。
ORIGIN:记录核酸序列,并以双斜线作为整条记录的结束符。
FASTA格式的核酸序列是最常用的序列书写格式。它由两部分组成,第一部分就是第一行,以大于号开始。大于号后面接序列的名称或注释。第二部分就是第二行以后的纯序列部分,这部分只能写序列,不能有其他内容,比如空格,注释,行号之类的都不能在序列部分出现。
FASTA格式:
>X01714.1 E. coli dut gene for dUTPase (EC 3.6.1.23) (deoxyuridine 5'-triphosphate nucleotidohydrolase)
CAGAGAAAATCAAAAAGCAGGCCACGCAGGGTGATGAATTAACAATAAAAATGGTTAAAAACCCCGATAT
CGTCGCAGGCGTTGCCGCACTAAAAGACCATCGACCCTACGTCGTTGGATTTGCCGCCGAAACAAATAAT
GTGGAAGAATACGCCCGGCAAAAACGTATCCGTAAAAACCTTGATCTGATCTGCGCGAACGATGTTTCCC
AGCCAACTCAAGGATTTAACAGCGACAACAACGCATTACACCTTTTCTGGCAGGACGGAGATAAAGTCTT
ACCGCTTGAGCGCAAAGAGCTCCTTGGCCAATTATTACTCGACGAGATCGTGACCCGTTATGATGAAAAA
AATCGACGTTAAGATTCTGGACCCGCGCGTTGGGAAGGAATTTCCGCTCCCGACTTATGCCACCTCTGGC
TCTGCCGGACTTGACCTGCGTGCCTGTCTCAACGACGCCGTAGAACTGGCTCCGGGTGACACTACGCTGG
TTCCGACCGGGCTGGCGATTCATATTGCCGATCCTTCACTGGCGGCAATGATGCTGCCGCGCTCCGGATT
GGGACATAAGCACGGTATCGTGCTTGGTAACCTGGTAGGATTGATCGATTCTGACTATCAGGGCCAGTTG
ATGATTTCCGTGTGGAACCGTGGTCAGGACAGCTTCACCATTCAACCTGGCGAACGCATCGCCCAGATGA
TTTTTGTTCCGGTAGTACAGGCTGAATTTAATCTGGTGGAAGATTTCGACGCCACCGACCGCGGTGAAGG
CGGCTTTGGTCACTCTGGTCGTCAGTAACACATACGCATCCGAATAACGTCATAACATAGCCGCAAACAT
TTCGTTTGCGGTCATAGCGTGGGTGCCGCCTGGCAAGTGCTTATTTTCAGGGGTATTTTGTAACATGGCA
GAAAAACAAACTGCGAAAAGGAACCGTCGCGAGGAAATACTTCAGTCTCTGGCGCTGATGCTGGAATCCA
GCGATGGAAGCCAACGTATCACGACGGCAAAACTGGCCGCCTCTGTCGGCGTTTCCGAAGCGGCACTGTA
TCGCCACTTCCCCAGTAAGACCCGCATGTTCGATAGCCTGATTGAGTTTATCGAAGATAGCCTGATTACT
CGCATCAACCTGATTCTGAAAGATGAGAAAGACACCACAGCGCGCCTGCGTCTGATTGTGTTGCTGCTTC
TCGGTTTTGGTGAGCGTAATCCTGGCCTGACCCGCATCCTCACTGGTCATGCGCTAATGTTTGAACAGGA
TCGCCTGCAAGGGCGCATCAACCAGCTGTTCGAGCGTATTGAAGCGCAGCTGCGCCAGGTATTGCGTGAA
AAGAGAATGCGTGAGGGTGAAGGTTACACCACCGATGAAACCCTGCTGGCAAGCCAGATCCTGGCCTTCT
GTGAAGGTATGCTGTCACGTTTTGTCCGCAGCGAATTTAAATACCGCCCGACGGATGATTTTGACGCCCG
CTGGCCGCTAATTGCGGCCAGTTGCAGTAATATGACGCCGGATGACTTTTCATCCGGCGAGTTTCTTTAA
ACGCCAAACTCTTCGCGATAGGCCTTAACCGCCGCCAGATGTTCCGCCATTTCCGGCTTCTCTTCCAGG
真核生物核酸序列
接着我们输入序号U90223
Title:Human deoxyuridine triphosphate nucleotidohydrolase precursor mRNA, nuclear gene encoding mitochondrial protein, complete cds
LOCUS HSU90223 960 bp mRNA linear PRI 03-JAN-1998
DEFINITION Human deoxyuridine triphosphate nucleotidohydrolase precursor mRNA,
nuclear gene encoding mitochondrial protein, complete cds.
ACCESSION U90223
VERSION U90223.1
KEYWORDS .
SOURCE Homo sapiens (human)
ORGANISM [Homo sapiens](https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=9606)
Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi;
Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini;
Catarrhini; Hominidae; Homo.
REFERENCE 1 (bases 1 to 960)
AUTHORS Ladner,R.D. and Caradonna,S.J.
TITLE The Human dUTPase Gene Encodes Both Nuclear and Mitochondrial
Isoforms: Differential Expression of the Isoforms and
Characterization of a cDNA Encoding the Mitochondrial Species
JOURNAL Unpublished
REFERENCE 2 (bases 1 to 960)
AUTHORS Ladner,R.D. and Caradonna,S.J.
TITLE Direct Submission
JOURNAL Submitted (19-FEB-1997) Dept. of Molecular Biology, Univ. of Med.
and Dent. of NJ-School of Osteopathic Medicine, 2 Medical Center
Drive, Stratford, NJ 08084, USA
FEATURES Location/Qualifiers
source 1..960
/organism="Homo sapiens"
/mol_type="mRNA"
/db_xref="taxon:[9606](https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=9606)" [CDS](https://www.ncbi.nlm.nih.gov/nuccore/U90223.1?from=63&to=821) 63..821
/note="mitochondrial dUTPase isoform; DUT-M"
/codon_start=1
/product="deoxyuridine triphosphate nucleotidohydrolase
precursor"
/protein_id="[AAB94642.1](https://www.ncbi.nlm.nih.gov/protein/2735292)"
/translation="MTPLCPRPALCYHFLTSLLRSAMQNARGTAEGRSRGTLRARPAP
RPPAAQHGIPRPLSSAGRLSQGCRGASTVGAAGWKGELPKAGGSPAPGPETPAISPSK
RARPAEVGGMQLRFARLSEHATAPTRGSARAAGYDLYSAYDYTIPPMEKAVVKTDIQI
ALPSGCYGRVAPRSGLAAKHFIDVGAGVIDEDYRGNVGVVLFNFGKEKFEVKKGDRIA
QLICERIFYPEIEEVQALDDTERGSGGFGSTGKN" [sig_peptide](https://www.ncbi.nlm.nih.gov/protein/AAB94642.1?from=1&to=69) 63..269
/note="mitochondrial targeting presequence" [mat_peptide](https://www.ncbi.nlm.nih.gov/protein/AAB94642.1?from=70&to=252) 270..818
/product="deoxyuridine triphosphate nucleotidohydrolase"
ORIGIN
1 ggtggaagcc tggcgcacgt ccggaggtgc cgaggaccca accagcccaa actctggggg
61 aaatgactcc cctctgccct cgccccgcgc tctgctacca tttccttacg tctctgcttc
121 gctcagcgat gcaaaacgcg cgaggcacgg cagagggccg aagccgcggt actctccggg
181 ccaggcccgc ccctcggccg ccggcggcgc agcacgggat tccccggccg ctgtccagcg
241 ctggccgcct gagccaaggc tgccgcggag ccagtacagt cggggccgct ggctggaagg
301 gcgagcttcc taaggcgggg ggaagcccgg cgccggggcc ggagacaccc gccatttcac
361 ccagtaagcg ggcccggcct gcggaggtgg gcggcatgca gctccgcttt gcccggctct
421 ccgagcacgc cacggccccc acccggggct ccgcgcgcgc cgcgggctac gacctgtaca
481 gtgcctatga ttacacaata ccacctatgg agaaagctgt tgtgaaaacg gacattcaga
541 tagcgctccc ttctgggtgt tatggaagag tggctccacg gtcaggcttg gctgcaaaac
601 actttattga tgtaggagct ggtgtcatag atgaagatta tagaggaaat gttggtgttg
661 tactgtttaa ttttggcaaa gaaaagtttg aagtcaaaaa aggtgatcga attgcacagc
721 tcatttgcga acggattttt tatccagaaa tagaagaagt tcaagccttg gatgacaccg
781 aaaggggttc aggaggtttt ggttccactg gaaagaatta aaatttatgc caagaacaga
841 aaacaagaag tcataccttt ttcttaaaaa aaaaaaaagt ttttgcttca agtgttttgg
901 tgttttgcac ttctgtaaac ttactagctt taccttctaa aagtactgca ttttttactt
//
由于真核生物的基因是非线性排列的,所以真核生物的数据库记录比原核生物的要复杂。这里我们选取了编码人dUTPase的成熟mRNA序列,其中已经剪切掉了内含子,只剩下外显子的序列,显得简洁。
在这里我们需要注意两点:
KEYWORDS:后面只有一个点。这个点提示我们,数据库并不是完美的,所有数据库都存在数据不完整的问题。JOURNAL:后面我们看到是写的是未正式发表。但事实上,这篇文章早在1997年就已经发表在JBC上了。
因此,忠言逆耳:别指望Genbank或任何一个数据库能够百分百做到数据无误且实时更新。
Features:里的注释内容与原核生物的数据库记录相似。
CDS:指出了从63到821是一段编码区,在这段编码区里基因是连续的,因为是经过剪切后的成熟RNA,它将被翻译成线粒体型dUTPase蛋白。
/translation:里给出的是计算机翻译出的该蛋白的序列。
sig_peptide:指出了编码信号肽的碱基的位置。信号肽决定了蛋白质的亚细胞定位,也就是蛋白质工作的地方。
mat_peptide:指出了编码成熟肽链的碱基的位置。
基因组数据库
我们查看人的基因组数据库(攻击33亿个碱基分布在23个染色体上)。
Ensembl genome browser 114是由欧洲生物信息学研究所EBI和英国桑格研究所合作开发的。它收入了各种动物的基因组。
微生物宏基因组数据库
JCVI Home Page | J. Craig Venter Institute是“人类微生物组计划”所建立的微生物数据库。
二级核酸数据库
二级核酸数据库包括的内容非常多。其中NCBI下属的三个数据库经常会用到。
RefSeq数据库:参考序列数据库,是通过自动及人工精选出的非冗余数据库,包括基因组序列、转录序列和蛋白质序列。RefSeq: NCBI Reference Sequence Database
dbEST数据库:也就是表达序列标签数据库,存储的是不同物种的表达序列标签。What is dbEST?
Gene数据库:以基因为记录对象为用户提供基因序列注释和检索服务,收录了来自5300多个扬种的430万条基因记录。Home - Gene - NCBI
一级蛋白质数据库
一级蛋白质数据库可以分为蛋白质序列数据库和蛋白质结构数据库。
蛋白质序列数据库
UniProt:合并了Swiss-Port、TrEMBL和PIR。
UniProt数据库有三个层次。
- 第一层叫
UniParc,收录了所有UniProt数据库子库中的蛋白质序列,量大,粗糙。 - 第二层是
UniRef,他归纳了UniProt几个主要数据库并且是将重复序列去除后的数据库。 - 第三层是
UniProtKB,他有详细注释并与其他数据库有链接,分为UniProtKB下的Swiss-Prot和UniProtKB下的TrEMBL数据库。
蛋白质结构数据库
蛋白质的结构可以分为四级:
- 一级结构:氨基酸序列
- 二级结构:α螺旋和β折叠
- 三级结构:蛋白质的三维空间
- 四级结构:几个蛋白质分子的复合体结构
以3H6X为例。
PDB文件解读
- 第一部分:头信息
HEADER:蛋白质结构的基本信息描述,包括分子类别,存储日期,PDB ID。
TITLE:结构的标题。
COMPND:对结构中各个分子的描述。从这里可以看出3H6X这个结构是由三条链形成的三聚体结构。
SOURCE:结构中所包括的每一个分子的实验来源。
KEYWDS:用于数据库搜索的关键词。
EXPDTA:测定结构所采用的实验方法。PDB中绝大部分结构都是通过X射线衍射法测定的,少数是核磁共振法,极少数是使用包括电子显微镜在内的其他方法测定的。
AUTHOR:作者信息。
REVDAT:历史上曾经对该数据库记录进行过的修改。
JRNL:发表结构的文献信息。
REMARK:无法归入其他部分的注释。
- 第二部分:一级结构信息(也就是氨基酸序列)
DBREF:该蛋白质在蛋白质序列数据库里的检索号等信息。
SEORES:氨基酸序列。
MODRES:对标准残基上的修饰,比如第56号位置的蛋氨酸被硒代蛋氨酸所取代。
- 第三部分:非标准残基信息
HET:非标准残基及位置。
HETNAM:非标准残基的化学名称。
FORMUL:非标准残基的化学式。
- 第四部分:二级结构信息
HELIX:位于螺旋结构上的氨基酸所在位置及所属链。
SHEET:位于折片结构上的氨基酸所在位置及所属链。
TURN:位于转角结构上的氨基酸所在位置及所属链。
Link:残基间的化学键。比如106号氨基酸上的C与107号氨基酸上的N之间的化学键是肽键!键长1.32埃。除了肽键还可能有氢键,二硫键等等。
- 第五部分:实验参数信息
CRYST1:晶胞参数。
ORIGXn:直角-PDB坐标。
SCALE*:直角部分结晶学坐标。
- 3D坐标信息
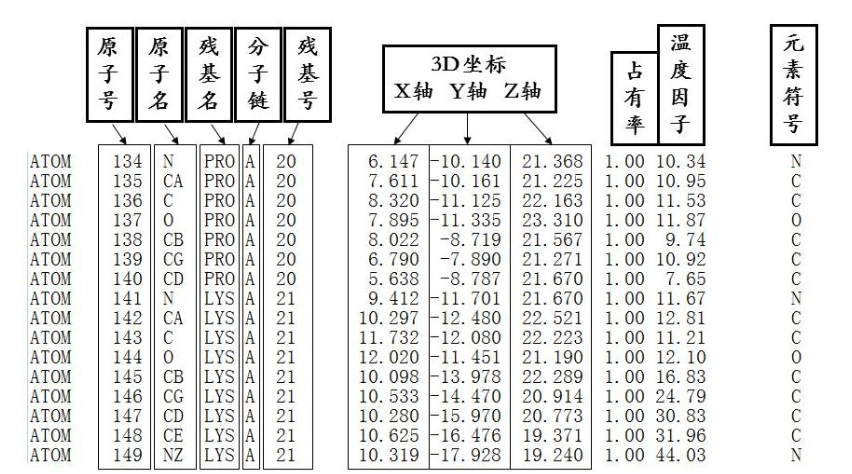
二级蛋白质数据库
蛋白质一般是由一个或多个功能区域组成,这些功能区域通常称作结构域(domain)。
在不同的蛋白质中结构域以不同的组合出现,形成了蛋白质的多样性。
Pfam is now hosted by InterPro数据库是一个蛋白质结构域家族的集合。
CATH: Protein Structure Classification Database at UCL蛋白质结构分类数据库。
SCOP| Structural Classification of Proteins蛋白质结构分类数据库。